空间单细胞测序技术|Nature—小鼠肝脏功能的区带模式
肝脏是一种由肝细胞组成的异质性组织,在维持代谢平衡过程中起到重要作用。在称为肝小叶的重复性结构单位中,肝细胞行使多种功能【1】。

肝小叶示意图【2】
六边形的肝小叶约由15个同心层组成【3,4】。血液从肝小叶边缘的肝门静脉和肝动脉流向中央静脉,构建了梯度变化的氧气、营养物质和激素微环境。此外,中央静脉分泌的Wnt形成素产生了相反的极化区域【5】。
与这一梯度变化微环境相一致的是,不同的同心层有着不同的职能,这是一种称之为“肝脏区带”的现象【6,7】。这种空间分工被认为使肝功能达到了最佳。例如,肝小叶外层高溶氧的肝门静脉中,参与耗能任务如糖异生和尿素生成的酶含量更高,而内层的中央静脉处专门进行糖酵解和外源物质代谢【8】。
事实上,前人已经使用RNA原位杂交和免疫组化技术研究了肝脏功能的区带模式【7,9】,确定了门静脉和中央静脉处肝细胞富集表达的基因不同。此外,单细胞测序技术的发展也使得研究者们得以认识上千个单细胞的基因表达水平【10,11】。
但由于这些技术自身的局限性,如灵敏度、通量和空间分辨度,当时仍缺乏在基因组水平上对肝脏区带现象的整体认识。
为此,Weizmann科学研究所的研究者们使用单分子荧光原位杂交(smFISH)和单细胞RNA测序(scRNA-seq)技术,精确地检测肝小叶不同坐标处分布着的mRNA,在基因组水平上重建肝脏区带模式。
该研究成果发表于2017年Nature。

影响因子:43.070
第一作者:Weizmann科学研究所的Keren Bahar Halpern博士
通讯作者:Weizmann科学研究所的Ido Amit教授以及Shalev Itzkovitz教授
开放数据:研究者对1,736个小鼠肝脏细胞进行的单细胞测序结果存储于NCBI,编号GSE84498。

为了在基因组水平上构建肝脏的区带模式,研究者选择了6个有着不同区带模式,且在肝脏高表达的基因:肝小叶中央附近的基因Glu1和Cyp2e1【12】,以及门静脉周围基因Ass1【13】、Asl10【13】、Alb8【7】和Cyp2f2【12】。
通过使用特定的核酸探针,研究者对禁食小鼠肝小叶轴向(肝门静脉—>中央静脉)上的肝细胞进行了smFISH分析(图1a),构建了肝细胞内特定基因的表达水平与该细胞所处位置间的概率关系。
根据smFISH分析结果获得的6个标志基因的区带模式谱,研究者将肝小叶轴向(肝门静脉—中央静脉)上的肝细胞划分成9个层(图1b)。

图1 肝小叶内不同空间位置上,标志基因的表达水平【21】。a是6个标志基因的smFISH显微图,其中灰色点是单个mRNA,红色是鬼笔环肽染色的膜;b是6个标志基因的区带模式谱。x轴是到中央静脉(CV)的成比例的距离,y轴是最大标准化表达水平,PN指的是肝门静脉。

研究者还使用MARS-seq技术,对超过1,500个的肝细胞进行了转录组测序,发现:
✪ t-SNE降维聚类分析结果揭示肝脏中存在3个不同的细胞聚群,分别是Kupffer细胞、内皮细胞以及肝细胞(图2a);
✪ 肝细胞表现出高度异质性的表达模式以及明显的梯度变化,这与6个标志基因的区带模式谱相似(图1b,图2b)。

a

b
图2 其中,a是t-SNE降维聚类分析结果,每个点代表着一个细胞,颜色分别表示的是肝细胞、Kupffer细胞以及内皮细胞的多个标志基因的表达水平;b是标志基因Cyp2e1和Cyp2f2的表达水平【21】。
为了确定单个细胞的空间位置,研究者开发了一种新的概率推断算法,根据单个细胞内6个标志基因的表达水平估算其空间位置,发现:
✪ 估算精确度极大地依赖标志基因在空间上的差异表达程度,层内细胞间基因组表达水平差异的影响较小(图3a);
✪ 对20个额外的、表达模式不同的基因进行smFISH分析,发现这两个图谱之间存在较好的整体相关性,确定了使用单细胞数据重建区带模式谱的准确度(图3b);
✪ 约50%的肝脏中表达基因在空间上具有区带模式(3,496个基因),这一基因数量比以前估测的要更多【12】(图3c);
✪ 比较单细胞数据以及前人数据中肝门静脉-中央静脉间的基因表达水平比值【12,13】,发现两者具有相关性(图3d)。

a

b

c

d
图3 其中,a是不同标志基因对区带模式谱的贡献【21】,x轴是每个基因表达水平标准化后的熵值,y轴是平均每一层的表达水平变异系数。b是使用20个额外基因验证重建的单个肝细胞区带模式谱的准确度(蓝色线是预测的区带模式谱中,该基因的表达模式;红色的是smFISH验证结果)。c右侧是3,496个呈显著区带模式基因的表达谱(右),蓝色表示的是低表达,黄色代表高表达;左侧是中央静脉(CV)周边、肝门静脉(PN)周边以及非单调谱基因的表达谱。d是前人研究结果与该研究的单细胞数据中,肝门静脉-中央静脉处基因表达水平的比值关系(左图【12】,右图【13】)。

可扩散的Wnt信号起源于中央静脉周边的内皮细胞,被认为在诱导中央静脉周边区带模式的形成中具有重要意义【5,8】。此外,有研究认为Ras信号诱导了肝门静脉周边区带模式的形成【14】。
因而,研究者分析了Wnt/Ras信号通路与空间位置的相关性,发现:
✪ 肝Wnt信号高活性的Apc-KO小鼠肝脏中【15】,表达上调的区带模式基因主要集中于中央静脉周边。这些被Wnt信号激活的基因中,有95个基因在慢性缺氧情况下出现表达上调,且明显偏向于在低氧含量的中央静脉层位置处表达(图4a、4b);
✪ 肝Wnt高活性的Apc-KO小鼠肝脏中【15】,表达下调的基因主要位于肝门静脉周边(图4a)。
这些结果表明Wnt信号和低氧含量是诱导中央静脉周边区带模式产生的主要因素。

a

b
图4 不同空间位置处,具有区带模式基因的表达水平【21】。其中,a是肝Wnt高活性的Apc-KO小鼠肝脏中【15】,被Wnt激活(绿色)或抑制基因(红色)的区带模式谱。b是在缺氧条件下被激活的95个基因(绿色)或被抑制的45个基因(红色)的区带模式谱。
✪ Ha-ras高活性肿瘤中【16】,与表达下调的区带模式基因相比,表达上调的区带模式基因明显倾向于在门静脉周边表达(图5a);
✪ 垂体机能减退的侏儒小鼠肝脏中,中央静脉周边的基因表达上调【17】,证明垂体激素抑制中央静脉周边基因的表达(图5b);
✪ 约2/3的区带模式基因不与Wnt、缺氧、Ras信号或垂体激素相关,意味着这些基因的区带模式受这些因子的联合调控,或受其他尚未被确定的形成素、血液因子调控。

a

b
图5 其中,a是Ha-ras高活性肿瘤中【16】,Ras激活的基因(绿色)以及Ras抑制的基因(红色)在不同空间位置处的表达水平;b是垂体机能减退侏儒小鼠的中央静脉周边【17】,表达上调(绿色)及下调(红色)基因的区带模式谱【21】。

为了探究生物学通路的空间分布模式,研究者对具有区带模式的3,496个基因进行了富集分析,发现:
✪ 富集了25个通路,其中氧化磷酸化通路在肝门静脉周边位置处表达水平更高,而该位置处的氧气、补体和凝血级联通路相关的分泌蛋白浓度较高(图6a);
✪ 编码肝脏分泌蛋白的基因倾向于在肝门静脉周边表达(图6b),这可能是一种更有效的分配方式,因为需ATP的血浆蛋白生成过程会消耗肝脏中约20%的氧气【18】;
✪ 倾向于富集在中央静脉周边的通路包括肝脏解毒途径(图6c),如外源物质代谢和谷胱甘肽代谢、胆汁酸生物合成以及蛋白酶体组分(图6a)。

a
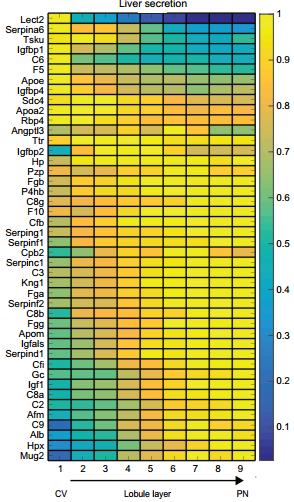 b
b c
c
图6 区带模式基因的KEGG通路富集分析结果【21】。a是不同空间位置上富集的KEGG通路;b是不同空间位置上富集的肝脏分泌蛋白;c是不同空间位置上富集的肝脏解毒基因。

前人描述的肝脏区带模式都是沿肝小叶的肝门静脉-中央静脉轴向呈单调递增或单调递减,而研究者构建的高分辨度的空间模式图谱展现了一种新的区带模式——在肝小叶中间层处达到峰值的非单调区带模式(图3c)。
进一步分析,发现:
✪ 通路富集分析结果没有揭示出非单调变化区带模式基因所富集的通路,但这些基因中包含关键的肝脏基因,例如编码铁调素(一种肝脏分泌的激素,能调控系统中铁元素水平)的Hamp及Hamp2(图7a);
✪ 多倍体肝细胞的非单调分布模式【19】与这些基因的非单调表达模式不存在相关性(图7b);
✪ Igf1在肝门静脉周边层中表达水平高,而其结合蛋白Igfbp2在肝小叶中部表达,结合蛋白Igfbp1在中央静脉周边层处表达。这一空间顺序可能代表着一种反馈系统,即Ifgbp的产生与上游分泌的Igf相匹配,或与Igfbp的其他非内分泌功能相关(图3b、图7c)。

a

b

c

d
图7 非单调区带模式及其随后的顺序【21】。a是Hamp的smFISH分析结果;b表明观察到的非单调区带模式谱与多倍体肝细胞的非单调分布模式无关,其中每个点代表着临近区域处不同倍数的一对细胞,其特定基因表达水平的Wilcoxon符号秩检验结果;c是Igfbp2的smFISH分析结果(上)和肝小叶不同层处的基因表达水平(下)。蓝色框内属于门静脉周边层、绿色框内属于肝小叶中部,洋红色框内属于中央静脉周边层。
此外,胆汁酸生物合成的中性途径核心顺序是Cyp7a1→Hsd3b7→Cyp8b1→Cyp27a1【20】。在使用单细胞数据构建的区带模式谱中,也显示出一致的基因表达顺序(图8):
✪ 中央静脉层1处Cyp7a1和Hsd3b7的表达水平最高,且该处不存在Baat基因的表达(该基因产生的酶能在胆汁酸分泌前与其发生共轭结合);
✪ Cyp8b1表达水平在肝小叶层2-3处达到峰值,1层处表达水平相对较低的原因可能是层1和层2间存在胆汁酸中间体的转移;
✪ 这种表达顺序还可能是因为Cyp7a1和Hsd3b7影响了肝小叶层1处的其他基因表达,或Cyp8b1的表达影响了层2处的其他基因表达。

a

b
图8 胆汁酸合成相关基因的空间表达模式【21】。其中,a是经典胆汁酸生物合成级联反应中,酶编码基因的空间表达顺序(上)以及肝小叶层2处的smFISH分析结果(下);b是Cyp8b1蛋白的免疫组化分析结果,与层1处(点线)相比,该蛋白质在层2-3处(虚线)的浓度更高。
总之,通过分析单细胞数据构建的区带模式表达谱,研究者确定了肝脏中存在区带模式分布的成对肝酶,这些酶在空间上的排列顺序明显不一致:这些不同位置处表达的酶要么作用于相同的底物,产生相同的产物(Ifgbp);要么依次作用,其中一种酶的产物是另一种酶的底物(胆汁酸生物合成过程)。

传统上,将肝细胞划分成肝门静脉周边和中央静脉周边两类肝细胞群,而研究者揭示了肝小叶内广泛的异质性,这为研究肝脏功能的空间分工模式及其背后的调控机制提供了基础,也为在组织水平上完整重建代谢结构提供了重要参考。

六边形肝小叶由洋葱样的肝细胞同心层组成。通过绘制一个肝小叶中所有肝细胞的基因活性谱,Weizmann的分子细胞生物系博士Shalev Itzkovitz及其研究团队将肝小叶分成9个层,揭示了每个层行使的功能不同。Itzkovitz说到:“我们发现肝细胞至少可以分成9种类型,每种专门行使的任务不同。”【22】
研究者发现,如葡萄糖、血栓因子和多种其他物质的合成发生在肝小叶的外层。“这些层中富含氧气,用以支持这些耗氧的合成过程。”Itzkovitz解释道。
肝小叶的内层处是破坏毒素和其他物质的场所。中层也被证实有其自身的功能,而不是仅作为过渡带。例如,研究者发现这些中层位置处肝细胞产生的激素,如铁调素,能调控血液中的铁元素水平。
研究者也发现特定过程,如胆汁的产生,需要经过经几个不同层的处理,就像是在一条生产线上。
总之,研究者发现肝脏中超3,500个基因的表达存在从一个层到另一个层的活性差异,这一数量约是以前估测的10倍【22】。
这种深度的基因表达分析或能帮助阐述常见肝脏疾病的过程和起源,包括肝癌和非酒精性脂肪肝疾病。这些疾病影响了发展中国家约五分之一的人口【22】。
绘真医学RedRockTM超高通量单细胞转录组测序分析,为您的研究提供有力支持!
1.SHALEV ITZKOVITZ LAB. (2019). "Single Cell Heterogeneity in the Mammalian Liver" http://shalevlab.weizmann.ac.il/research/
2. "Lobules of liver".Wikipedia.https://en.wikipedia.org/wiki/Lobules_of_liver
3.Hoehme, Stefan, et al. (2010). "Prediction and validation of cell alignment along microvessels as order principle to restore tissue architecture in liver regeneration." Proceedings of the National Academy of Sciences 107(23): 10371-10376.
4.Colnot, S. & Perret, C. (2011). "Molecular Pathology of Liver Diseases" Springer US (ed. Monga, S. P. S.):7-16.
5.Wang, Bruce, et al. (2015). "Self-renewing diploid Axin2+ cells fuel homeostatic renewal of the liver." Nature 524(7564): 180.
6.Jungermann, K. & Thomas K. (1996). "Zonation of parenchymal and nonparenchymal metabolism in liver." Annual review of nutrition 16(1): 179-203.
7.Gebhardt, Rolf, and Madlen Matz-Soja. (2014). "Liver zonation: Novel aspects of its regulation and its impact on homeostasis." World journal of gastroenterology: WJG 20(26): 8491.
8.Benhamouche, Samira, et al. (2006). "Apc tumor suppressor gene is the "zonation-keeper" of mouse liver." Developmental cell 10(6) : 759-770.
9.Gebhardt, Rolf. (1992). "Metabolic zonation of the liver: regulation and implications for liver function." Pharmacology & therapeutics 53(3): 275-354.
10.Zeisel, Amit, et al. (2015). "Cell types in the mouse cortex and hippocampus revealed by single-cell RNA-seq." Science 347(6226): 1138-1142.
11.Grün, Dominic, et al. (2015). "Single-cell messenger RNA sequencing reveals rare intestinal cell types." Nature 525(7568): 251.
12.Braeuning, Albert, et al. (2006). "Differential gene expression in periportal and perivenous mouse hepatocytes." The FEBS journal 273(22): 5051-5061.
13.Saito, Kosuke, Masahiko Negishi & E. James Squires. (2013). "Sexual dimorphisms in zonal gene expression in mouse liver." Biochemical and biophysical research communications 436(4): 730-735.
14.Hailfinger, Stephan, et al. (2006). "Zonal gene expression in murine liver: lessons from tumors." Hepatology 43(3): 407-414.
15.Gougelet, Angélique, et al. (2014). "T-cell factor 4 and β-catenin chromatin occupancies pattern zonal liver metabolism in mice." Hepatology 59(6): 2344-2357.
16.Unterberger, Elif B., et al. (2014). "Ha-ras and β-catenin oncoproteins orchestrate metabolic programs in mouse liver tumors." International journal of cancer 135(7): 1574-1585.
17.Boylston, W. H., James H. DeFord, and John Papaconstantinou. (2006). "Identification of longevity-associated genes in long-lived Snell and Ames dwarf mice." Age 28(2): 125.
18.Rolfe, D. F., and Guy C. Brown. (1997). "Cellular energy utilization and molecular origin of standard metabolic rate in mammals." Physiological reviews 77(3): 731-758.
19.Tanami, Sivan, et al. (2017). "Dynamic zonation of liver polyploidy." Cell and tissue research 368(2) : 405-410.
20.de Aguiar Vallim, Thomas Q., Elizabeth J. Tarling, and Peter A. Edwards. (2013). "Pleiotropic roles of bile acids in metabolism." Cell metabolism 17(5): 657-669.
21.Halpern, Keren Bahar, et al. (2017). "Single-cell spatial reconstruction reveals global division of labour in the mammalian liver." Nature 542(7641): 352.
22.(2017). ”Gene Analysis Adds Layers to Understanding How Our Livers Function” https://wis-wander.weizmann.ac.il/life-sciences/gene-analysis-adds-layers-understanding-how-our-livers-function.
更多信息请关注我们的网站
www.geno-truth.com/singlecell
联系电话:4000-672-118
服务邮箱:service@geno-truth.com

●文献解析|Science—scRNA-seq揭示水螅再生机制
文献解析|Nature—单细胞RNA测序首次揭秘人类胎肝造血作用(上)
文献解析|Nature—单细胞RNA测序首次揭秘人类胎肝造血作用(下)
单细胞空间转录组测序|Cell—小鼠原肠胚形成中期RNA空间模式






